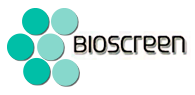结果
不同核酸提取方法对土壤原核生物多度与多样性的影响
与总DNA提取相比,DNase预消化和PMA处理均显著降低了土壤原核生物丰度,而碱性缓冲液洗涤和rRNA直接表征则没有观察到显著差异(图1A)。核酸提取方法对土壤原核生物丰富度和香农指数的分析也有显著影响(图1B、C)。
具体而言,与总DNA提取相比,DNase预消化导致原核生物丰富度显著下降了5.6%,而rRNA直接表征平均提高7.6%的扩增子序列变异(ASV)数(图1B)。尽管如此,与总DNA提取相比,碱性缓冲液洗涤和PMA处理未对原核生物丰富度产生显著影响(图1B)。不同核酸提取方法所表征的原核生物香农指数与丰富度的响应相似,但灵敏度更高(图1C)。除了总原核生物和活体原核生物之间的区别之外,在各个胞内核酸提取方法间,也观察到了活体原核生物多度和多样性的显著差异(图1A-C)。
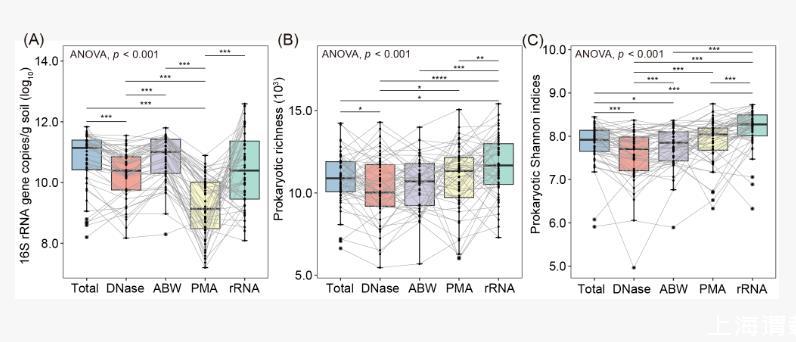
图1.不同核酸提取方法对土壤原核生物16S rRNA基因拷贝数、丰富度和香农指数的影响(A)16S rRNA基因拷贝数/克土壤(log10);(B)原核生物物种丰富度(103);(C)原核生物香农指数。Total:总DNA提取;DNase:DNase预消化;ABW:碱性缓冲液洗涤;PMA:叠氮溴化丙锭(PMA)处理;rRNA:rRNA直接表征。*p<0.05;**p<0.01;***p<0.001。
不同方法所表征土壤原核生物群落结构的差异
五种核酸提取方法共检测到62,939个共有ASV(占全部ASV的67.8%),共有ASV的平均相对多度为97.6%(图S1)。然而,不同方法所表征的多个原核生物类群的相对多度表现出显著差异(表S1;图2A–F及S2–6)。例如,与碱性缓冲液洗涤相比,DNase预消化显著降低了放线菌门的相对多度(图2A)。进一步分析表明,总体与活体原核生物的群落结构相似性仅约50%,其中碱性缓冲液洗涤法的相似性最高,而rRNA直接表征最低(图S5)。
此外,活体原核生物群落间的结构相似性为47.8%~66.2%(图S5)。总体与活体原核生物群落的相似性与多个环境因子显著相关,但其相关性的强度和性质因方法而异(图S7)。具体而言,基于DNase预消化法与总DNA提取所表征群落结构的相似性与土壤水分、总磷、总氮、总有机碳及黏粒含量呈显著负相关,而与砂粒含量呈正相关(图S7);rRNA直接表征与总DNA提取所表征群落结构的相似性与土壤总磷、总钾及总有机碳含量呈正相关,而碱性缓冲液洗涤和PMA处理所表征群落结构的相似性与环境因子相关性较弱(图S7)。
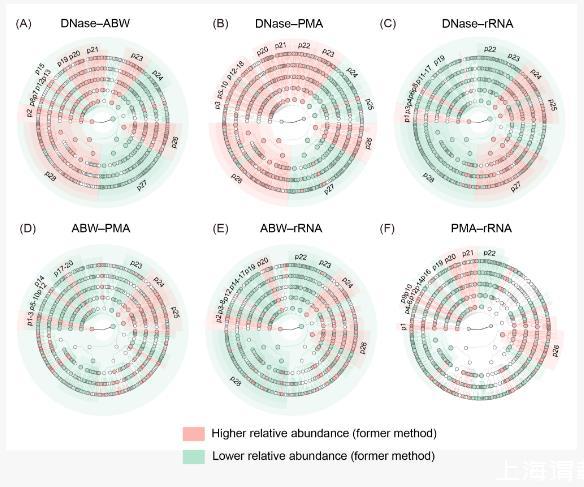
图2.不同土壤活体微生物研究方法所表征土壤原核生物类群的相对多度差异(A)DNase与ABW的比较;(B)DNase与PMA的比较;(C)DNase与rRNA的比较;(D)ABW与PMA的比较;(E)ABW与rRNA的比较;(F)PMA与rRNA的比较。
统计比较的详细说明见表S1。DNase:DNase预消化;ABW:碱性缓冲液洗涤;PMA:叠氮溴化丙锭(PMA)处理;rRNA:rRNA直接表征。红色节点表示前者相对多度明显高于后者的类群,绿色节点表示前者相对多度明显低于后者的类群。仅显示差异显著的原核生物类群。从内到外的环代表原核生物从界到属的分类级别。
不同方法所表征的土壤原核生物共现网络
不同方法所表征的土壤原核生物共现网络复杂度具有显著差异(图3A–E)。去除胞外DNA干扰后,网络复杂度显著降低:负连接数减少85.3%~98.5%,网络连通性下降15.6%~63.7%(表S2;图3A–E),但模块化程度提升了9.9%~47.8%(表S2)。总原核生物群落的网络鲁棒性显著低于活体原核生物(图3F),其鲁棒性排序为:rRNA直接表征>碱性缓冲液洗涤>PMA处理>DNase预消化>总DNA提取(图3F)。不同方法的网络模块中心节点(Module hubs)数量与组成差异显著,基于rRNA直接表征的网络模块中心点数量显著高于其他方法(图3A–E),且活体原核生物网络中41.7%~72.7%的模块中心点发生了更替(图S8)。
图3.基于不同核酸提取方法所表征的土壤原核生物共现模式(A–E)
基于不同核酸提取方法的土壤原核生物群落共现模式;(F)基于不同核酸提取方法的网络鲁棒性。根据分类信息对节点进行了着色,文本中的节点代表模块中心点。网络的鲁棒性是根据从网络中移除的节点比例所引起自然连通性的变化来评估的,自然连通性的下降率越高,表明网络的稳定性越低。Total:总DNA提取;DNase:DNase预消化;ABW:碱性缓冲液洗涤;PMA:叠氮溴化丙锭(PMA)处理;rRNA:rRNA直接表征。
不同方法所表征土壤原核群落构建机制的一致性
不同方法解析的原核生物群落结构与地理距离或环境因素有的关系十分相似(图4A和S9)。具体而言,随着地理距离的增加,原核生物群落结构的相似性显著下降(图4A)。
最主要区别是,基于rRNA直接表征的原核生物群落结构相似性的衰减速率远低于其他方法(图4A)。同样,不同核酸提取方法所表征的原核生物群落结构一般与年均温(MAT)、年均降雨量(MAP)、海拔、pH值、总氮(TN)和粉粒含量(图S9和表S3)显示出显著和相似的相关性。为进一步揭示环境因素对不同方法所表征原核生物群落结构的直接和间接影响,我们进行了结构方程模型分析。结果表明,地理位置、气候变量、土壤性质和质地共同解释了原核生物群落结构的大部分变异,
不同方法的解释方差略有不同:DNase预消化(74%)、碱性缓冲液洗涤(72%)、PMA处理(69%)、总DNA(69%)和rRNA直接表征(50%)(图S10)。此外,不同方法所揭示的群落构建机制高度一致(图4A–D)。修正随机比(MST)分析表明,随机过程和确定性过程在决定土壤原核生物群落构建方面几乎同等重要(图4B)。系统发生学(iCAMP)分析表明,同质性选择(HoS)、扩散限制(DL)和漂变是驱动土壤原核生物群落构建的主要过程,其贡献率分别为38.36%~43.5%、28.8%~31.1%和20.38%~24.04%(图4C)。相反,异质选择(HeS)和同质扩散(HD)的贡献几乎可以忽略不计(图4C)。中性模型(NCM)可解释原核生物群落结构中约75%的变异,且不同核酸提取方法所表征原核生物群落的Nm值相似(图4D)。
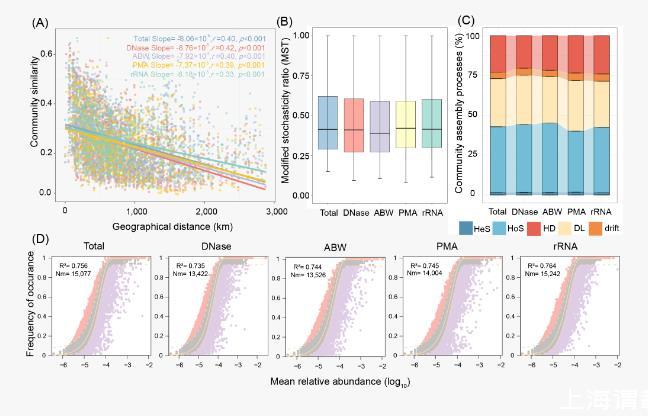
图4.基于不同方法的原核生物群落相似性距离衰减模式和构建机制(A)距离衰减关系;(B)基于修正随机比(MST)的群落构建机制;(C)基于iCAMP的群落构建机制;以及(D)中性群落模型(NCM)的适合度。HoS:同质选择;HeS:异质选择;HD:同质扩散;DL:扩散限制;Total:总DNA提取;DNase:DNase预消化;ABW:碱性缓冲液洗涤;PMA:叠氮溴化丙锭(PMA)处理;rRNA:rRNA直接表征。
相关新闻推荐
3、UL56基因下游重组病毒对鸭肠炎病毒生物特性、生长曲线影响——结果、结论