图2、新短小单胞菌菌株CHPC 1.3453T的进化关系。(A)24个短小单胞菌物种菌株的核心和附属基因组系统发育。每个物种的颜色对应于图1C中分支I到V的颜色。(B)CHPC 1.3453T与图1C泛基因组聚类分支V中密切相关成员的MAUVE比较。(C)在新菌株CHPC 1.3453T和分支V中其他密切相关成员中发现的共享和独特基因的维恩图。
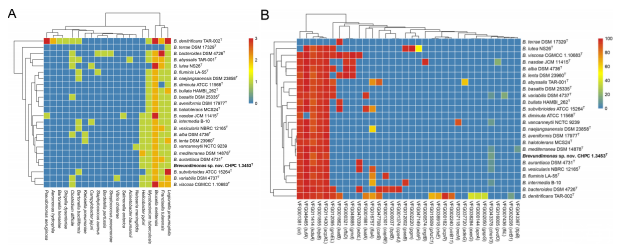
图3、短小单胞菌基因组中毒力相关基因的分布。(A)24个短小单胞菌物种关于毒力基因来源的层次聚类热图。从蓝色到红色的颜色代表毒力基因同源性相关频率。(B)24个短小单胞菌物种中毒力相关基因的层次聚类热图。颜色代表毒力相关基因序列的BLAST覆盖度值。
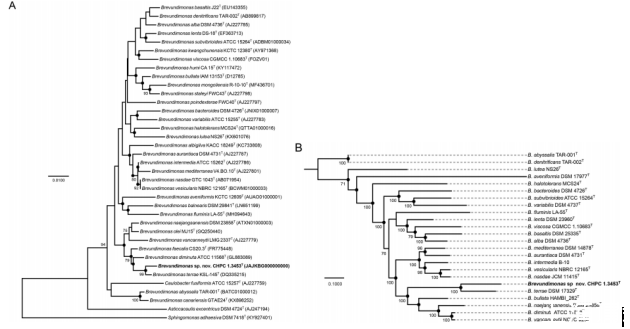
图4、短小单胞菌物种的系统发育树。(A)基于菌株CHPC 1.3453T和其他短小单胞菌物种分离株的16S rRNA基因序列构建的邻接树。基于1,000次重采样的自举值低于70%的未在分支节点显示。节点处的实心黑点表示通过邻接法和最大似然法同步恢复的属级分支。16S rRNA基因序列的GenBank登录号在括号中给出。三个菌株,即梭状柄杆菌ATCC 15257T、偏心中线菌DSM 4724T和粘固鞘氨醇单胞菌DSM 7418T,分别作为外群。水平条代表每个核苷酸位点0.01的替换。(B)基于5个看家基因串联序列(按gyrB-ppsA-recN-rpoC-rpoD顺序)构建的最大似然树。节点处的数字表示大于70%的自举值(1,000次重复的百分比)。实心黑点表示也通过邻接法和最大似然法恢复的属级分支。水平条代表每个核苷酸位点0.1的替换。
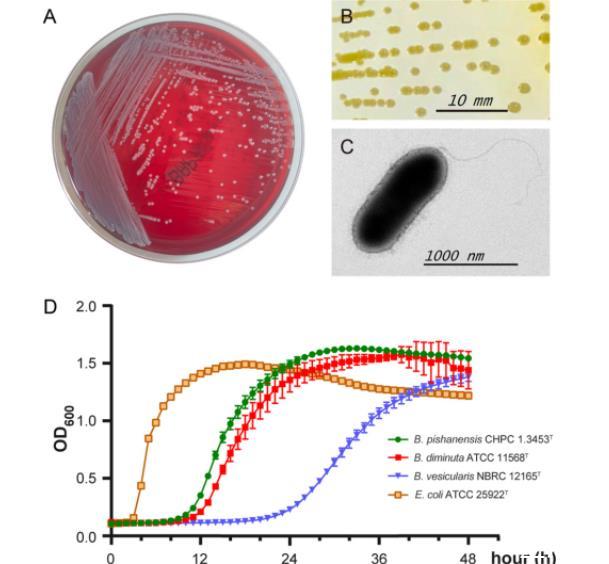
图5、菌株CHPC 1.3453T的培养和形态特征。(A)该菌株在血琼脂培养基上于35℃培养18小时后的生长情况。(B)在LB培养基上于35℃培养72小时后单个菌落的形态。比例尺代表10毫米的单位长度。(C)高分辨率透射电子显示的负染细菌细胞的一般形态。比例尺代表1000纳米的单位长度。(D)菌株CHPC 1.3453T的生长曲线。使用三个菌株,泡囊短小单胞菌NBRC 12165T、微小短小单胞菌ATCC 11568T和大肠杆菌ATCC 25922T作为对照。
总结
本论文旨在通过系统性的比较基因组学方法,深入探讨短小单胞菌属(Brevundimonas)的生物学特性、致病潜力及进化关系,并正式描述一个新物种——皮山短小单胞菌(Brevundimonas pishanensis sp.nov.)。研究团队首先从新疆皮山县分离获得一株具有典型慢生长特性的革兰氏阴性杆菌,通过16S rRNA基因序列分析初步确定其属于Brevundimonas属。为进一步确认其分类地位,研究人员对该菌株进行了全基因组测序,并与已知的Brevundimonas属多个代表菌株开展比较基因组分析。
结果表明,该菌株在平均核苷酸一致性(ANI)、数字DNA-DNA杂交(dDDH)值以及核心基因系统发育树上均显著区别于现有物种,符合新种界定标准,因此被命名为Brevundimonas pishanensis。此外,研究还聚焦于Brevundimonas属整体的致病性和生长特性。通过泛基因组分析发现,该属细菌普遍携带多种毒力因子相关基因,如外膜蛋白、铁摄取系统和分泌系统组件,提示其具备潜在致病能力,这与临床中偶有B.vesicularis引发败血症等感染病例的报道相吻合。同时,代谢通路注释显示,该属菌株普遍存在与能量代谢和氨基酸合成相关的基因缺失或简化,可能与其在自然环境中表现出的缓慢生长表型密切相关。研究还结合转录调控网络和蛋白质组分配模型,进一步解释了其生长速率受限的分子机制。
本研究不仅丰富了Brevundimonas属的物种多样性,为环境微生物资源库增添新成员,同时也从基因组层面揭示了该属细菌潜在的致病风险和生态适应策略,对临床感染防控、环境微生物监测及细菌进化研究具有重要参考价值。在本研究中,Bioscreen C自动浊度分析系统被用于定量监测细菌的生长动态,从而获取短小单胞菌属(Brevundimonas)菌株的生长曲线数据。