研究简介
基因组在细胞中的空间组织及其三维结构如何驱动细胞功能一直是生物学中的重大挑战。在细菌中,基因组DNA被压缩成一个高度有序、密集的状态,称为核样体(nucleoid)。尽管近年来在细菌三维基因组结构的研究上取得了一定进展,但由于Hi-C等方法的分辨率较低,核样体的精细结构和功能组织仍然不明确。在细菌中,基因组DNA被压缩成高度有序的浓缩状态,称为类核3,4,5。尽管近几十年来在表征细菌3D基因组结构方面取得了进展,但由于Hi-C 9,10,11等方法的低分辨率接触图,类核的精细结构和功能组织仍然难以捉摸。在这里,研究人员开发了一种增强的Micro-C染色体构象捕获,实现了10碱基对(bp)分辨率。这种超高分辨率分析揭示了大肠杆菌类核中的元素空间结构,包括染色体发夹(CHIN)和染色体发夹结构域(CHID)。这些结构由组蛋白样蛋白H-NS和StpA组织,在抑制水平转移基因方面发挥关键作用。H-NS的破坏会导致3D基因组的剧烈重组,减少CIN和CHID,而去除H-NS和StpA会导致它们完全分解、水平转移基因的转录增加和生长延迟。在netropsin上观察到类似的效果,它与H-NS和StpA竞争富含AT的DNA结合。CHIN之间的相互作用进一步将基因组组织成孤立的环,从而可能绝缘活性纵子。Micro-C分析表明,所有活跃转录的基因都以转录依赖性方式形成不同的纵子大小的染色体相互作用结构域(OPCID)。这些结构在Micro-C图上显示为方形图案,反映了整个转录区域的连续接触。这项工作揭示了大肠杆菌类核的基本结构元素,强调了它们与类核相关蛋白和转录机制的联系。
Bioscreen全自动生长曲线分析仪的应用
将大肠杆菌菌株的单个菌落在LB培养基中培养至固定期,然后用新鲜的LB培养基稀释至OD600约为0.08。培养物在37°C下生长,直到OD600达到约0.4。将每种培养物的150μl等分试样进一步稀释至约0.01的OD600,转移到蜂窝孔中,并使用Bioscreen C自动生长曲线分析仪在37°C下剧烈摇动孵育。每10分钟自动记录一次OD600值,并根据收集的数据绘制生长曲线。Bioscreen C自动生长分析仪在本研究中用于量化突变体(如Δhns/ΔstpA)和药物处理(如Rif、bleomycin)后的细菌生长表型。通过连续监测OD₆₀₀₀₀,对比野生型与突变体(如ΔmukBEF温度敏感株)的生长速率差异,为CHINs/CHIDs的结构稳定性提供生理学证据。
实验结果
开发一种增强型Micro-C染色体构象捕获技术,以10个碱基对的超高分辨率分析了大肠杆菌(Escherichia coli)基因组的三维(3D)结构,揭示了其基因组在活性和沉默状态下的基本组织形式。通过这种技术,分析了大肠杆菌基因组的三维结构,发现了几种基本的空间结构,包括染色体发夹(CHINs)和染色体发夹域(CHIDs)。这些结构由类组蛋白H-NS和StpA组织,对抑制水平转移基因(HTGs)的表达起关键作用。破坏H-NS会导致三维基因组的显著重组,减少CHINs和CHIDs,而同时去除H-NS和StpA则会导致它们完全解体,增加水平转移基因的转录,并导致生长延迟。H-NS缺失会导致3D基因组重组并减弱CHINs/CHIDs,而H-NS/StpA双敲除则完全破坏这些结构,伴随HTGs的转录激活和生长延迟。此外,转录活跃的基因形成操作子大小的染色体互作域(OPCIDs),其结构与转录活性直接相关,表现为Micro-C图谱中的方形接触模式。
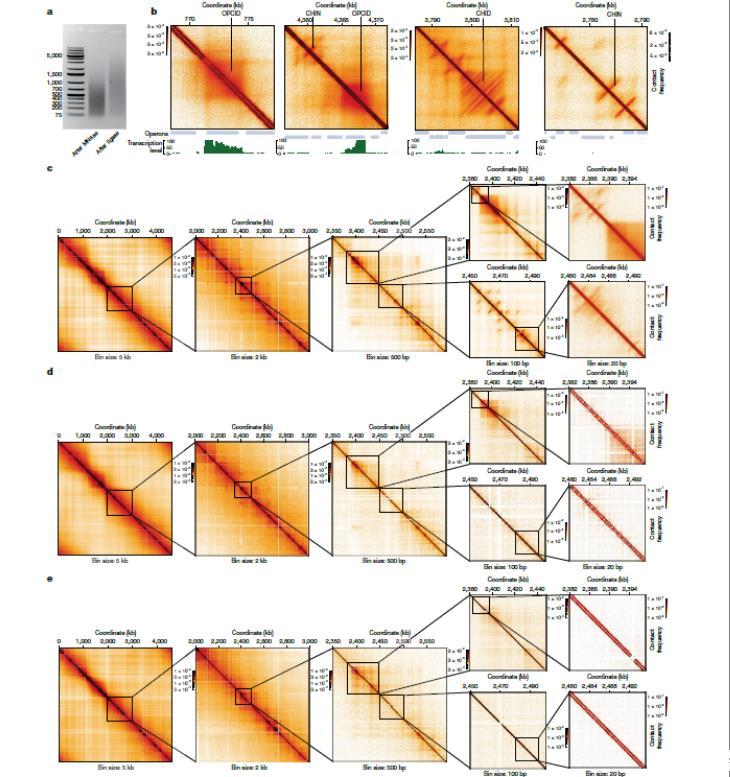
图1、超高分辨率Micro-C图谱揭示了大肠杆菌基因组的复杂3D组织。a)在Micro-C程序的不同步骤后对DNA进行琼脂糖凝胶电泳,展示了超高分辨率映射所需的片段化模式。b)Micro-C接触图示例,显示OPCID、CHIN和CHID以及转录活性谱和纵子轨迹。c-e,Micro-C和Hi-C数据比较:正常生长条件下WT大肠杆菌细胞生成的Micro-C图谱(c);使用标准Hi-C协议在相同细胞上生成的Hi-C接触图(d);大肠杆菌MG1655(e)的Hi-C图谱。
相关新闻推荐
2、猪流行性腹泻病毒生长曲线的测定及盐霉素对其抑制效果(五)
3、乳酸乳球菌F44生长曲线、pH曲线、酸耐受、Nisin耐受和Nisin效价(三)