2、结果与讨论
2.1植酸酶AppA靶向基因修饰系统构建
为了实现pGSA-appA基因在目标细菌基因组中16S rRNA基因位点的靶向修饰,需要对原始pSPIN载体骨架进行逐步改造。原始pSPIN的Spacer引导序列靶向定位于lacZ基因(图2A)。第一步,将原始Spacer序列改造为针对目标菌株的16s-spacer(该序列引导整个转座复合体对靶点的识别),见图2B。第二步,以16s-pSPIN为基础,对Cargo转座子的启动子进行改造。原始载体的Cargo基因缺少启动子,因此采用组成型启动子J23119,以实现目的基因的稳定表达,改造后得到质粒J23119-16s-pSPIN(图2C)。随后,在J23119-16s-pSPIN质粒基础上对Cargo转座子区域进行改造,将pGSA-AppA融合蛋白基因酶切(图3B)并连接在J23119启动子后10 bp左右处,构建最终的植酸酶AppA表面展示系统载体,使其能够在16S rRNA基因位点上表达(图2D)。
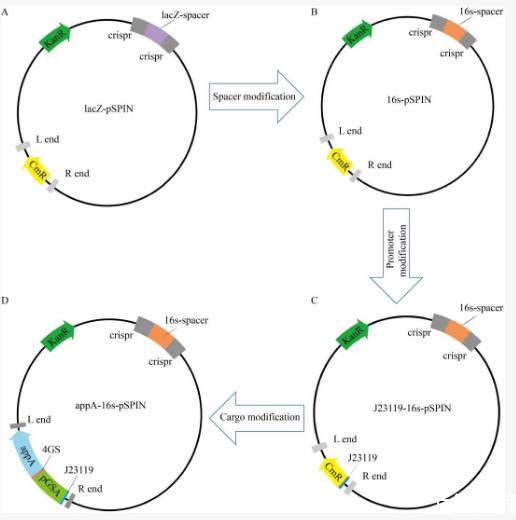
▲图2 pGSA-appA-pSPIN载体的构建流程
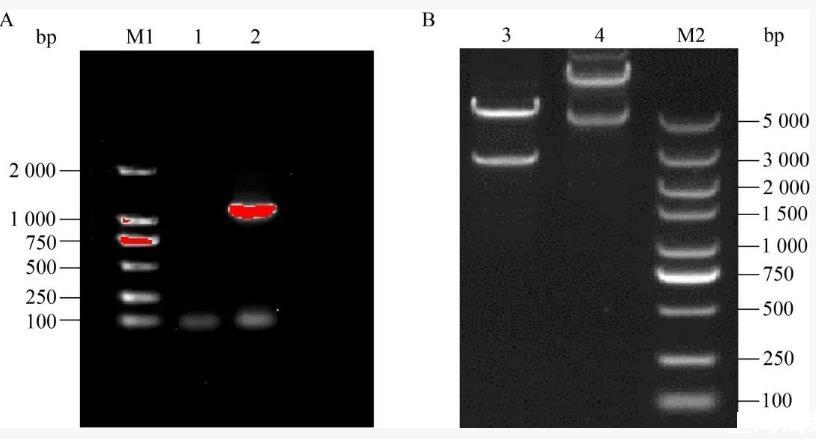
▲图3转座插入(A)和pGSA-appA载体构建(B)的PCR凝胶图谱。M1:DL2000 DNA marker;泳道1:土著菌;泳道2:基因修饰菌;3:pGSA-appA酶切验证;4:pET-30a-pGSA的载体克隆验证;M2:DL5000 DNA marker。
锚定蛋白pGSA是枯草芽孢杆菌的γ-谷氨酸合成酶A(poly-γ-glutamic synthetase A)膜蛋白,被广泛应用于蛋白质的表面展示。该锚定蛋白具有表面展示效率高、使用方便等优点,且表面展示后的蛋白具有更高的活性和环境耐受性。AppA植酸酶最初发现于大肠杆菌,由其基因组中的appA基因编码,属于组氨酸酸性磷酸酶(HAP)家族。植酸酶AppA的三维结构包含一个催化域(负责水解植酸)和一个结构辅助域(增强了底物结合能力),是一种典型的6-植酸酶(6-phytase),即优先从植酸的D-6位点开始水解磷酸酯键,依次生成IP5、IP4等低磷酸化肌醇衍生物,最终产物是无机磷酸和肌醇。
根据从黑土中分离的土著细菌皮氏罗尔斯顿氏菌(Ralstonia pickettii)G3的16S rRNA基因序列特征,设计pGSA-appA-pSPIN的Spacer序列,用于pGSA-appA的基因修饰,靶向位点位于27F后962 bp处。INTEGRATE系统通过Spacer序列的引导,会将整个R end-L end的Cargo区域转座至目标位点。为了验证转座插入的成功,针对插入序列设计了特异性引物(27F/CM2R),对嵌合16S rRNA基因的27F到R end之间的序列进行PCR,其中CM2R是R end上的特征序列。凝胶电泳结果显示,由于外源基因已成功插入基因组的目标位置形成融合基因,因此可以通过特异性引物完成PCR过程;而原始菌株由于缺乏Cargo转座区,因此无R end段序列,进而无PCR产物(图3A)。R.pickettii G3属于革兰氏阴性好氧菌,致病性较低,是环境中常见的一种细菌,能够适应复杂及恶劣环境,在许多环境污染修复方面表现出潜力,并且该菌可以通过共代谢的方式,与其他微生物互利共生,促进生长并提高环境修复效果。核糖体16S rRNA由细菌基因组上的基因序列转录而成,rrnDB数据库显示,Ralstonia通常含有3-4个16S rRNA基因拷贝,因此其冗余的16S rRNA基因可以作为基因修饰的位点。
INTEGRATE系统的PAM序列为“CC”,紧随其后的是引导的Spacer序列。对PCR测序结果显示,pGSA-appA的Cargo区域插入到Spacer序列后49 bp处(图4)。通常Cargo区域会插入到靶点Spacer序列后50 bp左右处,这与本研究的结果一致。Klompe等研究结果表明,当转座子序列的两端(L end和R end)交换位置后其转座效率更高。因此,本研究采用“R end”在前的载体构建方法(图1)。
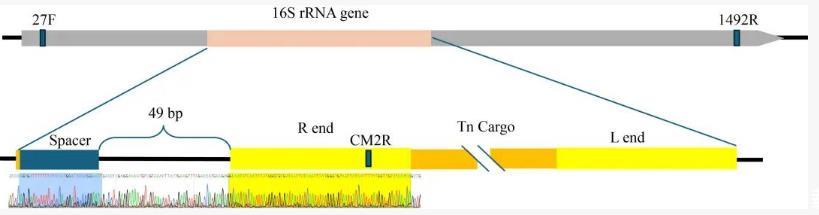
▲图4 pGSA-appA的插入位点
基因修饰是通过在宿主细胞内插入一段目的基因或删除某段特定基因,从而使原有宿主获得新的功能或改变其基因型。理想的基因修饰技术应不依赖DNA断裂及同源重组,同时具备特异性和可编程性。基于CRISPR技术的最新进展为微生物染色体的便捷整合提供了新的思路和方法。传统的基因工程菌构建通常使用模式菌株,或者具有特殊整合位点的菌株。最新研究表明,CRISPR-Cas系统可以与细菌类Tn7转座子结合,完成RNA引导的转座整合。随着该技术的不断改进,INTEGRATE编辑系统得以开发并初步应用。该系统主要包括3个部分:目标基因、TnsABC转座酶复合体以及Cas蛋白串,最终目标基因在向导RNA的引导下,在目标位点下游50 bp左右处完成整合。然而,将该技术应用于黑土根际微生物菌群的功能基因修饰方面,目前仍处于空白状态。目前,该系统的插入目标位点主要为LacZ以及一些经过筛选的安全位点,缺乏更便利的候选插入位点。本研究使用16S rRNA基因作为修饰基因的靶点,并验证了该位点的可行性,从而实现了在任意菌株上快速的靶向转座和基因修饰。值得注意的是,由于载体的转化需要借助E.coli WM3064的结合转移,因此该方法仅适用于革兰氏阴性菌。此外,更为复杂的环境,例如土壤中的微生物菌群能否实现快速的基因整合,也是重要的研究方向。
2.2基因修饰菌的生长和植酸酶活性检测
在生长曲线对比分析中发现,与未改造的原始菌相比,基因修饰菌在稳定期的OD600吸光度略低,但二者较为接近(图5A)。由于转座的位置是核糖体的16S rRNA基因位点,核糖体主要负责蛋白质的合成和加工,对其位置进行改造可能会导致细菌生长状况的改变。然而,本研究结果表明,对冗余的16S rRNA基因进行嵌入不会明显抑制或影响细菌的生长。
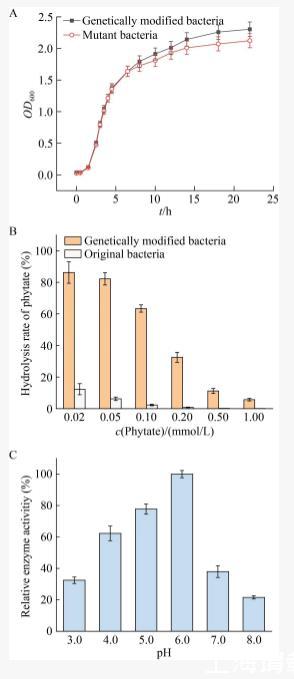
▲图5基因修饰菌的生长趋势对比及对植酸的水解。A:生长曲线对比;B:不同底物浓度的水解率;C:pH对植酸酶活性的影响。
随后,检测了植酸酶基因修饰菌在不同植酸浓度条件下的水解能力。结果显示基因修饰菌表现出更高的水解能力(图5B),其水解植酸的能力是原始菌的8倍以上。由于原始菌拥有其他种类的磷酸酶,这些酶也会在一定程度上水解植酸,因此原始菌也表现出一定的植酸水解能力,但远低于基因修饰菌。此外,在低浓度植酸条件下,基因修饰菌(OD600值为1.0)可以在1 h内水解超过80%的植酸,并且其水解率会随着底物浓度升高而降低。
pH对基因修饰菌植酸酶活性的影响如图5C所示。随着pH值的增加,酶活性呈现出先增加后降低的趋势,在pH 6.0时表现出最高的相对酶活性,随后在碱性环境下酶活性会迅速下降,其相对酶活性在pH 8.0时仅为20%左右。植酸酶是一种酸性磷酸酶,因此碱性环境不利于植酸酶活性的维持,之前的研究结果也表明植酸酶在碱性环境中活性会迅速降低。
为了使植酸酶在基因修饰菌中稳定表达,本研究使用了组成型的J23119启动子。该启动子可以不依赖诱导物而自行启动下游基因的表达,并且其表达量适中,基本不会影响宿主的生长。目前,只有少数植酸酶成功展示在细胞表面,且常用模式生物作为宿主,例如酵母和枯草芽孢杆菌,并且其表面展示通常借助表达载体。虽然表达载体使用较为便捷,但其使用会导致一些潜在问题,例如持续的筛选压力、抗生素抗性基因转移、载体丢失等,这使得其在使用过程中存在更高的风险。本研究将整个植酸酶表面展示的融合蛋白转座至细菌基因组,可以保证其稳定表达,且由于该系统是无痕编辑,因此不存在抗生素抗性基因泄漏的风险。
2.3土壤有机磷活化
在添加植酸酶基因修饰的R.pickettii G3菌后,处理组的土壤表现出更高的植酸酶活性,是对照土壤植酸酶活性的2倍以上。在第21天时,植酸酶活性缓慢下降,但其活性仍可长期保持,在28 d后其植酸酶活性依然保留1.5倍左右(图6A)。土壤植酸酶活性结果显示,植酸酶基因修饰菌株极大地提高了对土壤植酸的水解潜力。
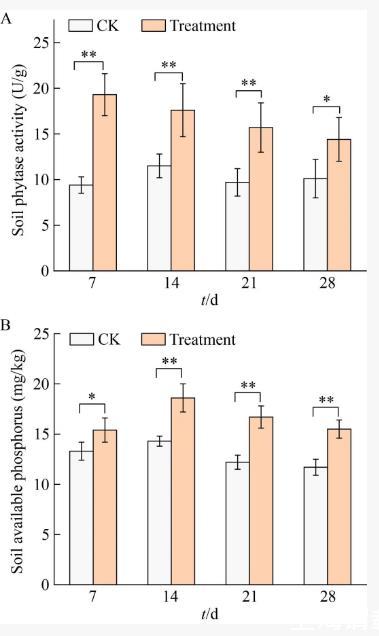
▲图6土壤植酸酶活性和有效磷含量变化。A:土壤植酸酶活性对比;B:土壤速效磷含量对比。
植酸酶会水解土壤中的植酸并释放磷酸根,从而提高土壤速效磷含量。对土壤速效磷的检测结果显示,从第7天开始,土壤速效磷含量显著提高,在第14天后达到最高水平,提高了近30%(图6B)。由于大豆对土壤磷的摄取,土壤中磷含量会缓慢下降,但整体上基因修饰菌添加组的速效磷含量高于对照组。其他研究结果表明,提高土壤植酸酶活性可以显著提高土壤速效磷水平,最高可提高54.48%的速效磷含量,并且这种促进效果可以维持较长时间。此外,其他研究也采用类似策略,通过添加外源植酸酶的方式,水解了土壤中近17.0%的植酸,并提高了22.5%的有效磷含量。目前,许多产植酸酶的根际菌被用于植物促生,接种促生菌后,根长、磷含量、生物量和产量均有显著改善。然而,野生菌的遗传调控通常难以控制,实际应用可能会超出预期。
转基因微生物在环境修复和药品生产等领域表现出巨大潜力。随着转基因技术的发展,转基因微生物的环境风险可以得到有效控制和评估。CRISPR基因组编辑是一种典型的“无标记”基因编辑技术,可避免抗性基因的潜在危害。此外,荷兰的研究团队进行了30多年的观察,未发现转基因细菌有明显的环境影响,表明它们是可以安全使用的。
3、结论
目前,已报道的CRISPR引导转座研究中均缺乏通用的基因转座位点。许多细菌基因是单拷贝基因,并且由于环境中的细菌通常为营养缺陷型,许多细菌并不一定拥有已报道的转座靶位点,例如LacZ基因,这无疑限制了INTEGRATE等CRISPR转座技术的应用。本研究基于CRISPR引导的转座插入技术,创造性地将目标菌的16S rRNA基因位点作为靶点,进行快速基因修饰。16S rRNA基因位点作为转座靶点,一方面省去了复杂的安全位点筛选,同时由于其为多拷贝基因,因此在基因组中部分16S rRNA基因位点的转座并不会对转基因菌的生长造成影响。此外,通过实验证明,功能基因可以快速完成靶向转座,其位置可以通过Spacer序列进行精确定位。此外,细胞分裂基因也是微生物所共有的基因之一,同样可以作为基因修饰的候选靶点。
完成外来基因在目标细菌基因组上的整合后,其功能能否发挥是另一个重要的研究内容。本研究使用组成型启动子J23119,可保证目的基因在土壤环境中的稳定表达。结果显示,经过植酸酶基因修饰的土著菌获得了极高的植酸酶活性,并且其功能具有较宽的pH谱。将基因修饰菌施用于黑土后,土壤植酸酶活性提高了2倍左右,土壤速效磷含量也显著提高。因此,通过CRISPR转座介导的基因修饰可以赋予土著菌新的功能。植酸酶活性的提高可以有效活化土壤有机磷库,释放速效磷。通过基因修饰的微生物策略对于黑土等土壤的元素活化具有一定的潜力和研究价值。