1、材料与方法
1.1菌株和基因
植酸酶appA基因(Uniprot ID:P07102)克隆自大肠杆菌K12,锚定蛋白pGSA基因(ACT52837.1)采用全基因合成(合成基因的载体骨架为pET-30a),锚定蛋白和植酸酶的融合可参考文献,融合后的载体为pET-30a-pGSA-appA。其中,锚定蛋白pGSA和乘客蛋白AppA之间需用柔性肽(GGGGSGGGGS)连接,以消除pGSA和AppA之间的干扰,融合后的表面展示蛋白命名为pGSA-appA。大肠杆菌(Escherichia coli)DH5α和一株从黑土中分离的菌株皮氏罗尔斯顿氏菌(Ralstonia pickettii)G3分别作为基因的克隆和表达宿主,质粒转化通过二氨基庚二酸(diaminopimelic acid,DAP)营养缺陷型的大肠杆菌(Escherichia coli)WM3064作为供体菌进行偶联转移实现。INTEGRATE系统所用载体pSPIN骨架的构建步骤可参考文献。其中,CRISPR间隔区的Spacer序列(32 nt)用于基因的靶向定位,R end和L end之间的区域为Cargo区(其中包含目的基因)。具体基因和引物序列见表1。

表1本研究使用的基因和引物序列
1.2主要试剂和仪器
卡那霉素,生工生物工程(上海)股份有限公司;二氨基庚二酸,Sigma-Aldrich公司;植酸钠、细菌基因组提取试剂盒、土壤速效磷含量检测试剂盒,北京索莱宝科技有限公司;超级感受态细菌制备试剂盒、Taq酶、Pfu酶,上海碧云天生物技术股份有限公司。
PCR仪,Bio-Rad公司;其他所需材料可参考文献。
1.3载体构建
本研究使用的最终表达载体可参考图1中的pGSA-appA-pSPIN,该载体主要由QCascade(CRISPR区、Cas基因表达区)、TnsABC(转座酶表达区)和Donor(Cargo区)3部分组成。载体构建的步骤包括Spacer区改造、启动子改造和Cargo区改造,其中植酸酶融合蛋白的启动子为J23119组成型启动子。利用SnapGene软件构建载体图谱,引物设计和无缝克隆模拟也通过该软件完成。载体的构建步骤主要包括载体线性化和无缝连接,详细步骤参考文献。
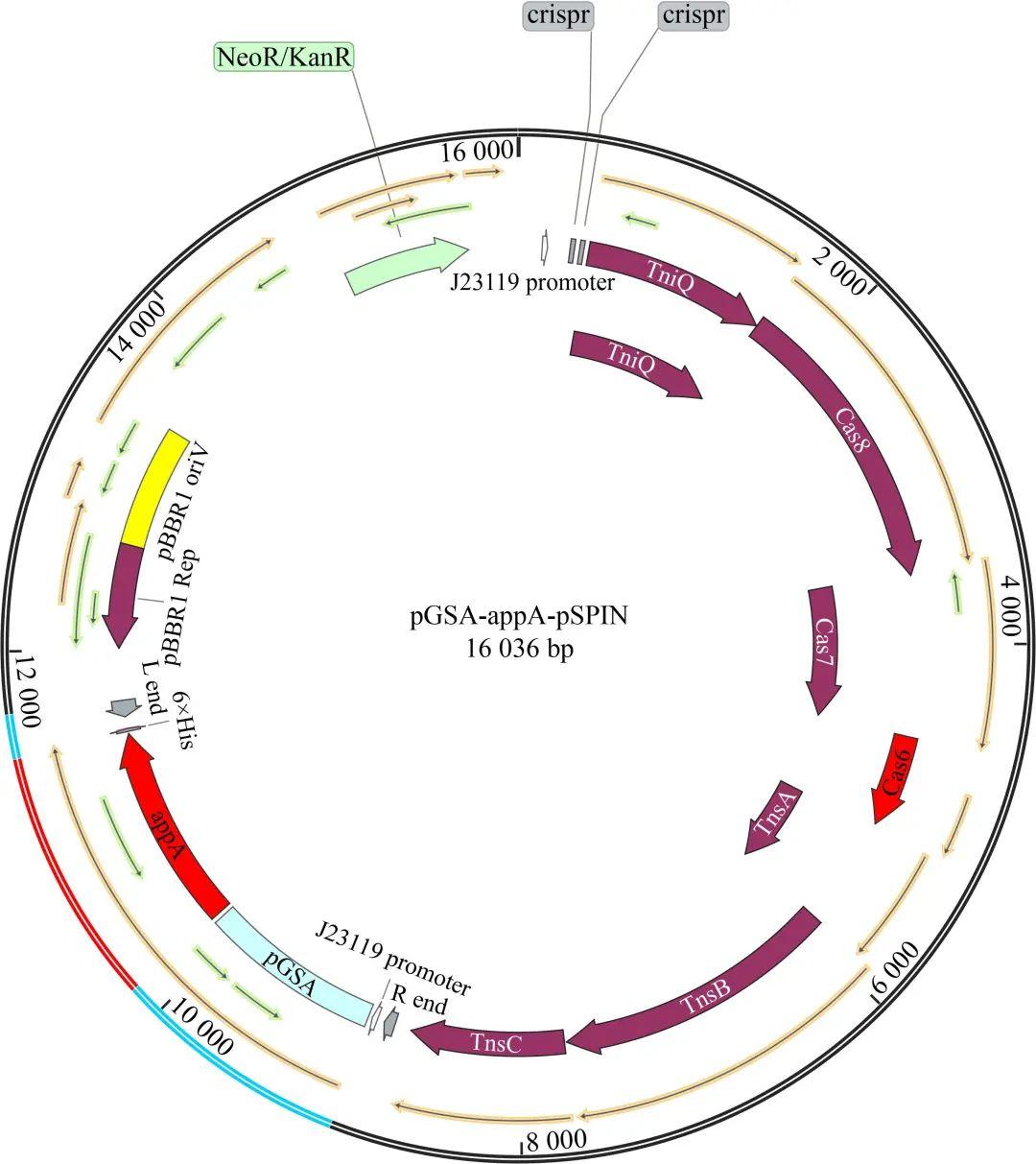
▲图1 pGSA-appA-pSPIN载体图谱
1.4载体的接合转移
使用E.coli DH5α扩增目的载体pSPIN-pGSA-appA,并通过超级感受态细菌制备试剂盒制备E.coli WM3064供体感受态,随后将pSPIN-pGSA-appA转化至E.coli WM3064。将携带载体的供体菌株WM3064与受体菌R.pickettii G3按照2:1的比例置于含有50μg/mL卡那霉素和50μmol/L DAP的LB培养基中,37℃培养过夜(~10 h),具体过程可参考文献。利用卡那霉素抗性筛选接合子后,使用细菌基因组提取试剂盒提取接合子基因组,并通过引物27F(5ʹ-AGAGTTTGATCCTGGCTCAG-3ʹ)和CM2R(5ʹ-CACCCATAAATTGATAATTATCACACCC-3ʹ)对接合子基因进行PCR,PCR反应可参考文献完成。
1.5植酸酶活性检测
植酸酶活性的测定采用改良的磷酸根显色法,该方法主要通过磷酸根与钼酸盐反应生成磷钼酸复合物,随后被还原生成蓝色钼蓝,最后根据样品的吸光度值在标准曲线中换算磷酸根浓度。
100 mmol/L的NaAc-HAc缓冲液:取4.101 5 g无水乙酸钠,溶于500 mL蒸馏水中,用乙酸调节至pH 6.0。植酸钠底物溶液:配制6.25 mmol/L的植酸钠溶液,取1.443 5 g肌醇六磷酸钠,溶于NaAc-HAc缓冲液中(现配现用)。终止液:5%三氯乙酸溶液(5%TCA)。显色液:将4份试剂A与1份试剂B混合,现配现用。试剂A:取7.5 g钼酸铵,溶于400 mL蒸馏水中,加入22 mL浓硫酸,定容至500 mL,配成1.5%的钼酸铵溶液,4℃保存。试剂B:配制2.7%硫酸亚铁溶液,取6.75 g十二水硫酸亚铁,溶于250 mL蒸馏水中,4℃保存。KH2PO4母液(50 mmol/L):取0.680 5 g KH2PO4固体,溶于100 mL 0.1 mol/L的NaAc-HAc缓冲液中。
取0.1 mL菌液,置于37℃水浴预热5 min,加入0.4 mL底物溶液并充分混合,置于37℃水浴反应30 min后,加入0.5 mL终止液及0.5 mL显色液,显色10 min,检测OD700数值。
取0.0、0.8、1.6、2.4、3.2、4.0 mmol/L的KH2PO4标准液各0.1 mL,置于37℃水浴预热5 min。反应结束后,取1 mL上述样品于狭缝比色皿,用分光光度计测量OD700,根据分光光度计数值绘制标准曲线。
1.6基因修饰菌的酶活性能分析
取对数生长期(OD600约为0.7)的基因修饰菌液,7 000 r/min离心5 min,去上清后收集菌体。菌体用生理盐水吹洗3次,将清洗后的菌体重悬于生理盐水中,制成细胞密度为1.0×108 CFU/mL的细胞悬浮液。
设置不同植酸钠底物浓度,取0.1 mL上述菌液于37℃水浴预热5 min;加入不同浓度的植酸钠底物溶液0.4 mL,混合均匀后置于37℃水浴反应30 min;加入终止液,混匀;加入显色剂显色10 min;取1 mL上述样品于狭缝比色皿,用分光光度计测量OD700;通过标准曲线获取水解后的磷酸根浓度。
取0.1 mL菌液于37℃水浴预热5 min,加入0.1 mmol/L的植酸钠底物溶液0.4 mL,并使用盐酸和氢氧化钠溶液分别调节pH至3.0、4.0、5.0、6.0、7.0、8.0,37℃水浴反应30 min,后续检测方法同上。
1.7基因修饰菌对土壤植酸的活化
将500 g过筛黑土土壤与10 mL生理盐水清洗后的R.pickettii G3菌液(OD600值为1.0)混合均匀后,放入5粒大小均匀的已发芽大豆种子,每组重复5次(以无菌的生理盐水作为对照)。从播种大豆开始,每7天取一次表层土壤,储存于-80℃冰箱待测。称取0.1 g过筛土样,加入1 mL NaAc-HAc缓冲液,1 500 r/min振荡1 h,10 000 r/min离心10 min,取上清液0.1 mL作为待测样品(空白组将菌液换成缓冲液即可),按照植酸酶活性检测方法进行操作。土壤植酸酶活性(单位U/g)计算如公式(1)所示。
土壤植酸酶活性(U/g)=[(A₁-A₀)×k×L×D]/(M×T×V)(1)
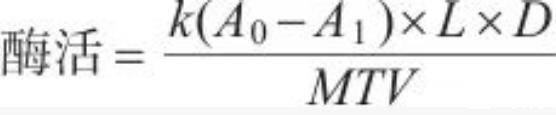
式中:k为标准曲线斜率,A1和A0分别为反应30 min和对照的OD700值,L为提取植酸酶时加入的缓冲液总体积,D为提取液稀释倍数,M为称取的土样质量,T为反应时间,V为反应液的体积。
取不同时间段的土壤,利用试剂盒检测土壤速效磷含量,检测方法可参考试剂盒说明书。使用SPSS 30.0.0进行ANOVA分析。
相关新闻推荐
1、细菌生长曲线图用于评估菌血症患者的大肠杆菌载量与疾病严重程度关联性