摘要胞外DNA在土壤中的长期留存已得到充分证实,但其对微生物多度与多样性分析的干扰却仍存争议。这一争议可能主要源于有关现有活体微生物研究方法可靠性的认知局限。本研究基于在中国西部地区所采集的土壤样品,系统比较了碱性缓冲液洗涤、叠氮溴化丙锭(PMA)处理、DNase预消化和rRNA直接表征等4种常用方法在表征土壤活体原核生物群落方面的表现。研究发现:胞外DNA的去除显著影响了原核生物多度、多样性、群落结构和共现网络的解析,但对群落构建机制解析的影响近乎可以忽略。不同研究方法所表现出的影响却同样显著差异:相比于基于总DNA的研究,DNase预消化与PMA处理显著降低了原核生物多度,基于碱性缓冲液洗涤与rRNA直接表征法的分析则未观察到显著差异;DNase预消化显著降低了物种丰富度,rRNA直接表征法则增加了原核生物丰富度。尽管67.8%的扩增子序列变体(ASVs)为共有类群,但基于不同方法所表征的各微生物类群的相对多度存在显著差异。此外,胞外DNA去除降低了共现网络的复杂性,却增强了网络的鲁棒性。DNase预消化展现出最高的DNA去除效率与活体微生物表征准确性,而其他方法却存在去除效率低、结果稳定性差或相关结果解释的不确定性高等问题。本研究表明不同方法所表征的活体原核生物群落存在显著差异,推荐使用DNase预消化法进行土壤活体原核群落研究,这为优化微生物组研究方法提供了重要指导。
引言
土壤微生物作为生物地球化学循环的核心驱动者,在维持生态系统功能与健康中发挥关键作用。当前的研究主要依赖于扩增子测序、宏基因组分析、基因芯片和定量PCR等技术。值得注意的是,源于裂解微生物的细胞外DNA可在土壤中持续存在数周至数年,占土壤总DNA库的近40%。其存在的时间受温度、湿度、pH和有机质等环境因子调控:低温与高有机质含量能够延缓降解,而高温高湿的环境则会加速分解。然而现行微生物分析多基于总DNA提取,其结果可能受胞外DNA的干扰。因此,消除胞外DNA的潜在影响成为当前土壤微生物组学研究的重要挑战。为应对这一挑战,研究人员开发了一系列方法来研究活体微生物,包括碱性缓冲液洗涤、叠氮溴化丙锭(PMA)处理、DNase预消化和rRNA直接表征等。其中碱性洗涤能有效去除胞外DNA,曾是研究活体微生物最常用的方法。
PMA在可见光下与胞外DNA形成不可逆的共价交联,从而抑制后续PCR扩增,而活体细胞凭借完整膜结构可排斥PMA进入,使其DNA得以提取和扩增,近年来PMA处理法在研究土壤活体微生物多度和多样性方面得到了广泛应用。DNase I作为去除胞外DNA的另一种有效方法,主要通过水解磷酸二酯键降解胞外DNA,也常被用于土壤活体微生物组研究。此外,因rRNA在胞内含量高,且在环境中极不稳定,已有学者提出,rRNA表征法也可用于研究活体微生物群落。总之,这些方法原理迥异,可能对活体微生物群落解析产生显著有影响。
事实上,关于胞外DNA对微生物多样性分析的影响,不同方法的研究结论存在矛盾。例如,基于PMA处理的多项研究都观察到胞外DNA显著影响了土壤微生物多样性。相比之下,一项基于DNase预消化法的研究则未观察到胞外DNA对土壤细菌多样性的显著影响。因此,比较和评估解析土壤活体微生物的常用方法是土壤微生物学领域的迫切需要。目前,已有一些研究初步评估了相关方法在研究土壤活体微生物多度和多样性方面的效率。然而,相关研究主要集中于基于纯培养菌株或针对有限数量环境样本的单一方法。
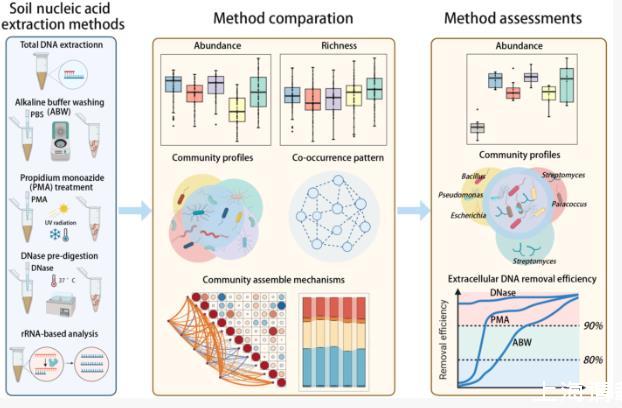
目前,尚未有基于大尺度环境样本对土壤活体微生物研究方法进行系统的比较和评估。因此,本研究基于在中国西部采集的土壤样本,系统地比较和评估了研究活体微生物群落的碱性缓冲液洗涤法、PMA处理法、DNase预消化法和rRNA直接表征法等4种常用方法。样点的海拔、年均温和年均降水量范围分别为21~4618 m、-6.04~21.83℃和40.7~1565.1 mm。测定并比较了基于不同方法所表征土壤原核生物的多度、多样性、共现模式和群落构建机制。
此外,还通过标签DNA添加实验评估了各方法的胞外DNA去除效率,并采用模拟群落验证了各方法在表征活体群落方面的可靠性。本研究重点验证了“不同方法在表征活体原核生物多度与多样性方面存在显著差异”这一核心假设。