使用相同的策略和补充表S1中列出的引物构建nrfA和cymA框内缺失突变体。使用cre-lox系统生成napB框内缺失突变体,引物列于补充表S1,产生△napB。分别使用融合PCR方法将pDS-NAPA和pDS-NRFA引入△napB突变体,构建双突变体△napA△napB和△napB△nrfA。
构建互补质粒并用作对照,以确保观察到的表型是由于基因缺失所致。所有用于PCR扩增的质粒和引物列于补充表S1。
在本研究中,厌氧生长测定在LB-1(胰蛋白胨10gl⁻¹,酵母提取物5gl⁻¹,NaCl 0.5gl⁻¹,乳酸钠20mM,pH=7.0)中进行,该培养基源自LB,并补充以下一种电子受体:NaNO₃(2mM)、NaNO₂(2 mM)、富马酸盐(20mM)、TMAO(20mM)、二甲亚砜(20mM)、MnO₂(5mM)和柠檬酸铁(10mM)。虽然LB-1培养基比定义的培养基M1、HEPES和MOPS支持更快的生长速率和更高的生物量,但本研究未观察到矛盾的结果。此外,补充了乳酸和电子受体的LB先前已用于S.oneidensis在厌氧条件下的生理表征。为避免离子色谱分析中Cl⁻的干扰,将NaCl的最终浓度降低至0.5%。在Bioscreen C微生物生长曲线分析仪中通过监测三次重复样品中OD600的增加来确定S.oneidensis菌株在厌氧条件下的生长。
对在硝酸盐与富马酸盐上生长的MR-1进行微阵列分析。在厌氧条件下,将50ml补充了2mM硝酸钠或10mM富马酸盐(对照)作为电子受体的LB-1接种至OD600为0.01,并在厌氧室内生长至对数中期(OD600≈0.15)。每种电子受体三个独立制备的培养物作为生物学重复,在Sorvall RC5C plus中以8000rpm离心3分钟(室温),立即将沉淀在液氮中冷冻并储存于-80°C。按照我们实验室的标准程序进行总RNA提取、cDNA标记、杂交和载玻片扫描。
使用LOWESS对数据集进行归一化,随后应用方差分析(ANOVA)和Benjamini and Hochberg错误发现率作为多重检验校正进行统计分析。原始微阵列数据已存入基因表达综合数据库(GEO),登录号为GSE11198。除了显示两个样品之间表达差异的常规双色微阵列分析外,还计算了绝对表达值(信号强度)以确定每个样品中的绝对RNA水平。在这种情况下,对所有重复中每个基因的信号强度进行统计分析,因为数据来自单染料微阵列杂交。
通过离子色谱法测定硝酸盐、亚硝酸盐和铵的浓度。在不同时间点收集培养样品,用0.2μm过滤器过滤并进行离子色谱分析。使用配备IonPac AS19柱的ICS-3000测定培养物中的硝酸盐和亚硝酸盐浓度,使用配备IonPac CS12柱的ICS-1000测定铵浓度。使用的洗脱液分别为浓度为100 mM、流速0.6 ml min⁻¹的Na₂SO₄(用于ICS-3000)和浓度为20mM、流速0.6ml min⁻¹的甲磺酸(用于ICS-1000)。
测定完整细胞中硝酸盐还原为亚硝酸盐和亚硝酸盐还原为铵的速率。收集在补充20mM富马酸盐的LB-1中厌氧生长至对数中期(OD600≈0.25)的细胞,离心收集,用新鲜LB-1培养基洗涤两次,并重悬于LB-1中至OD600约为0.2。取200μl等分试样,使用二喹啉甲酸测定试剂盒,以牛血清白蛋白为标准,按照制造商的说明测定蛋白质浓度。通过向测定培养基中加入2mM NaNO₃或NaNO₂来启动硝酸盐/亚硝酸盐还原反应。每隔15分钟取200μl等分试样,持续2小时,用0.2μm过滤器过滤,并进行上述离子色谱分析,测定硝酸盐、亚硝酸盐和/或铵。通过比较添加底物的消失速率和/或相应产物的出现速率,计算完整细胞将硝酸盐转化为亚硝酸盐和亚硝酸盐转化为铵的速率。
在厌氧条件下液体培养基中的竞争实验。为准备野生型和△napB菌株之间竞争实验的接种物,将每种菌株的厌氧培养物在补充2mM硝酸钠的LB-1中独立生长至稳定期(OD600约0.2)。将每种培养物各5ml混合,作为T0样品,并将同一混合物的100μl接种到9.9ml新鲜的补充2mM硝酸盐的LB-1中。培养24小时后,将100μl竞争细胞接种到新鲜的9.9ml相同培养基中,其余部分作为T1样品。第二天重复实验,收集样品作为T2。总共连续重复该程序5天。所有样品用新鲜LB系列稀释,将100μl适当稀释的样品铺在LB平板上。从含有150-300个菌落的平板上随机挑取100个菌落,使用补充表S1中列出的引物进行菌落PCR。相对适应度W根据先前描述的方法计算。
结果
对S.oneidensis nap和nrf基因的微阵列分析。S.oneidensis基因组中蛋白质编码基因的注释随时间发生了变化。根据原始注释,S.oneidensis拥有由操纵子napDAGHB(SO0845-9)和基因napF(SO1663)编码的NAP用于硝酸盐还原。在亚硝酸盐还原方面,注释显示存在许多NRF基因,包括nrfA(SO3980)、nrfF(SO0477)、nrfGCD(SO0482-4)和nrfD-2(SO4568)。在最新的注释中,nrf基因发生了显著变化,而nap基因保持不变。基于新的转录谱数据,最新的注释将nrfF和nrfGCD分别与c型细胞色素生物发生和硫代硫酸盐还原相关联。除了将nrfD-2(SO4568)重命名为nrfD外,该注释还将染色体上nrfD(SO4568)旁边的SO4570和SO4569指定为nrfB和nrfC。然而,由于截断,两者都被提议为假基因。
为了深入了解硝酸盐还原中的基因,使用S.oneidensis全基因组cDNA微阵列进行了转录谱分析。进行了初步实验以评估硝酸盐和亚硝酸盐对S.oneidensis生长的毒性。在存在2mM硝酸盐的情况下,生长未受到明显影响(数据未显示)。尽管2 mM亚硝酸盐使生长起始延迟了约2小时,但使用无法支持可检测生长的较低浓度是不切实际的(数据未显示)。因此,在整个研究的所有实验中,我们向培养基中提供2 mM水平的硝酸盐和亚硝酸盐作为电子受体。将在厌氧条件下在2mM硝酸盐或10mM富马酸盐上生长的MR-1细胞在对数期取样进行分析。在取样点时,硝酸盐和亚硝酸盐的浓度分别约为0.3和1.7 mM。使用先前报道的方法对阵列数据进行统计学质量评估。在六个注释的nap基因(napDAGHB和napF)中,观察到操纵子napDAGHB所有成员的转录增加,而napF的转录未受影响(图1)。在nrf基因方面,我们的分析中观察到nrfA的转录显著增加,但nrfD及其截断的伙伴nrfC和nrfB未受影响,表明NrfD可能在亚硝酸盐还原过程中不活跃。
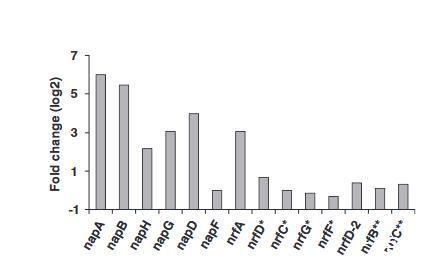
图1 通过微阵列分析显示的MR-1细胞在硝酸盐与富马酸盐培养条件下预测nap和nrf基因的转录水平。所用基因名称基于TIGR注释标准。带单星号标记的基因在最新注释中已更名,带双星号标记的基因已退化。
此外,硝酸盐的存在对nrfF(SO0477)和nrfGCD(SO0482-4)的转录影响很小,这些基因在原始注释中被预测参与硝酸盐/亚硝酸盐还原,但在最新注释中未提及。所有这些结果表明,关于硝酸盐还原中的基因,最新注释更可能是正确的。