NicA2和Pnao的表征
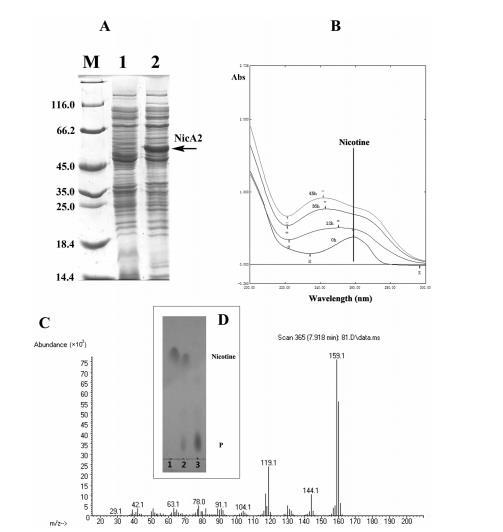
为了验证该新型基因的预测,从S16菌株克隆了nicA2并在大肠杆菌BL21(DE3)细胞中表达(文本S1)。IPTG诱导后,在大肠杆菌裂解物中发现了大约50 kDa的大量蛋白质,而在未诱导细胞或仅含有载体的细胞中观察不到这样的条带(图6A)。含有pET28a-nicA2质粒的静息细胞用于转化尼古丁,并且它们可以降解尼古丁,如UV扫描分析所示;因此证实nicA2基因产物具有预期的功能(图6B)。通过TLC和GC-MS分析鉴定了中间体N-甲基米斯明(P),并且我们观察到与先前报道相同的结果(图6C和图6D)[11]。
用Ni-NTA亲和柱纯化了His6标记的NicA2,并通过SDS-PAGE确认了酶的纯度,在接近50 kDa处观察到单一条带(图S5A)。进一步的实验证据支持FAD参与NicA2的功能。在382和452 nm处的紫外可见最大吸收峰,在410 nm处有最小吸收峰,是黄素蛋白的特征(图S5B)。分别敲除了nicA2和pnao基因,并进行了相关的细胞生长和静息细胞反应(文本S1)。nicA2和pnao基因缺失突变体不能在尼古丁培养基平板和液体培养物中生长(图7A和图7C)。
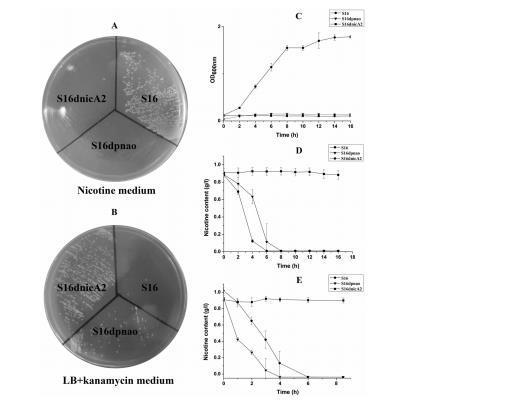
然而,缺失突变体S16dpnao在含有1 g l-1尼古丁的LB培养基中可以很好地降解尼古丁,其速率与野生型菌株S16相当(图7D)。与S16菌株相反,nicA2基因缺失突变体的静息细胞不能降解尼古丁(图7E)。pnao基因缺失突变体可以在含有1 gl-1尼古丁的LB培养基中生长并将其颜色变为深黄色,而nicA2缺失突变体的培养基颜色没有变化。此外,pnao基因缺失突变体的静息细胞降解尼古丁,但它不能进一步降解N-甲基米斯明,并且不能利用尼古丁作为唯一碳源和氮源(图7A、7C和7E)。上述事实表明酶NicA2和Pnao对于该菌株的尼古丁降解至关重要。
材料与方法
化学品
L-(-)-尼古丁(纯度99%)购自Fluka Chemie GmbH(Buchs Corp., Switzerland)。测序级胰蛋白酶来自Promega(Madison, Sweden)。SP(98%)来自Toronto(Canada)。所有其他试剂均为分析级并可商业获得。
细菌菌株、培养条件和测定
恶臭假单胞菌S16在含有1 g l-1尼古丁作为碳源和氮源的培养基中培养,如前所述[9]。该细菌也在含有1 g l-1甘油、1 g l-1 (NH4)2SO4和矿物盐培养基中培养,初始pH为7.0,含有13.3 g l-1 K2HPO4·3H2O、4 g l-1 KH2PO4、0.2 g l-1 MgSO4·7H2O和0.5 mg微量元素溶液。微量元素溶液(每升0.1 M HCl)包含以下材料和数量:0.05 g CaCl2·2H2O、0.05 g CuCl2·2H2O、0.008 g MnSO4·H2O、0.004 g FeSO4·7H2O、0.1 g ZnSO4、0.1 g Na2MoO4·2H2O和0.05 g Na2WO4·2H2O。尼古丁、SP和HSP的定量数据通过高效液相色谱(HPLC)分析获得,根据先前的报告[11,12]。
用于HPLC-MS/MS分析的蛋白质组制备
将在尼古丁或甘油作为碳源生长的恶臭假单胞菌S16细胞分别悬浮在PBS缓冲液中。用PBS缓冲液洗涤后,将细胞重悬在缓冲液(8 M尿素,0.05% SDS,10 mM DTT,10 mM Tris,pH 8.0)中,并用液氮研磨裂解。在12,000 g(10分钟,4°C)离心后,将上清液与丙酮(预冷)按体积比1:4混合。在-20°C过夜孵育后,将混合物在12,000 g(10分钟,4°C)离心。用预冷丙酮洗涤沉淀三次,并重悬在含有6 M Gu-HCl,100 mM Tris,pH 8.3的缓冲液中。使用改良的Bradford方案测定蛋白质含量。将酶溶液(100μg)悬浮在含有10 mM DTT的缓冲液中,在56°C下放置0.5小时,然后在25°C下加入50 mM IAA放置40分钟。经过3 K超滤膜超滤并用100 mM NH4HCO3冲洗膜后,将溶液的pH调节至8.0-8.5。将40μg测序级修饰胰蛋白酶加入提取物中,并在37°C下温和旋转(蛋白质:胰蛋白酶比例=50:1)进行消化过夜。
2D-LC/MS和蛋白质鉴定
为了从细胞混合物中鉴定蛋白质,使用了蛋白质组学中的多维液相色谱,使用1100 LC系统。第一维开始是用真实的连续线性盐梯度(0-130分钟,2%-35%;130-135分钟,35%-90%;135-140分钟,90%;140-141分钟,90%-2%;141-180分钟,2%)从硅胶强阳离子交换柱(0.075 mm x 5 cm)和C18柱(0.075 mm x 10 cm)(Column Technology Inc.)洗脱肽段。色谱条件:缓冲液A:H2O;缓冲液B:乙腈。纳喷雾柱直接连接到LTQ Classic离子阱质谱仪(Thermo Fisher)的孔口。纳喷雾电离通过3.5 kV的喷雾电压和200°C的加热毛细管温度实现。m/z范围从400到1800。
蛋白质组生物信息学
使用蛋白质组学发现软件1.2(ThermoFisher, CA, USA)对MS或MS/MS谱进行数据库搜索。在整个色谱运行过程中收集质谱图。通过Bioworks分析质谱图。自动识别和组织高得分肽段匹配。使用来自注释的恶臭假单胞菌S16基因组的蛋白质数据库,该数据库包含总蛋白质条目。假定电荷状态Z=1且XCorr得分>2.2,或电荷状态Z=3且XCorr得分>3.75的肽段匹配被自动接受为有效。
RNA提取和实时定量逆转录PCR(RT-qPCR)
从基本培养基平板上随机挑取恶臭假单胞菌S16的单个菌落,并将新鲜过夜培养物的1:100稀释液接种到三个250 ml锥形瓶中的50 ml基本培养基(对照)和添加了1 g l-1尼古丁的基本培养基(诱导)中。分批培养在30°C下以摇动(200 rpm)培养至中期指数期(OD600 1.4)。通过14,000 g离心2分钟收获中期指数期细胞,并将沉淀在-70°C储存过夜(16小时)。使用RNAprep pure细胞/细菌试剂盒(Tiangen)从约1x10^9个恶臭假单胞菌S16细胞中提取总RNA,并通过NanoVue(GE Healthcare)定量。使用随机六聚体引物和SuperScript III逆转录酶(Invitrogen)将0.8 mg经DNase(Fermentas)处理的总RNA逆转录为cDNA。将cDNA稀释1:10,并作为qPCR分析的模板,使用CFX96实时PCR检测系统(Bio-Rad)与SYBR Green RealMasterMix(Tiangen)和qPCR引物(表S4)。使用熔解曲线和琼脂糖凝胶分析确认PCR产物的特异性。用恶臭假单胞菌S16基因组DNA的十倍稀释液构建每个引物对的标准曲线。实验通常使用恶臭假单胞菌S16的对照和尼古丁诱导培养物进行三次重复,并将每个靶基因的阈值周期(CT)值归一化到参考基因16S rRNA基因。使用2^–ΔΔCT方法计算相对表达水平,其中ΔΔCT = (CT,靶标 - CT,16S)诱导 - (CT,靶标 - CT,16S)对照[30]。
构建spmABC基因破坏的突变体恶臭假单胞菌S16dspm
通过PCR从恶臭假单胞菌S16总DNA中扩增携带5'和3'截短的spmA片段,并克隆到pK18mob的多克隆位点中,pK18mob是一种不能在假单胞菌中复制的可移动质粒。PCR引物序列如下:spmA-Sall, CCACGTCGACCAAGTTAACTGGTTATGCGAC 和 spmA-EcoRI, CCACGAATTCAGTCCTTGGCCGAAACTTTGC。通过双亲滤膜接合[31]将重组质粒从广宿主范围动员菌株大肠杆菌S17-1转移到恶臭假单胞菌S16。供体和受体分别在37°C和30°C的LB肉汤中培养至OD600mm 0.6。将10^9个供体细胞和2x10^9个受体细胞用0.85% NaCl洗涤三次,混合并涂布在置于LB琼脂上的0.45-μm滤膜上。平板在37°C正面朝上培养5小时,然后在30°C培养12小时。然后将细胞重悬在0.5 ml 0.85% NaCl中,并以不同稀释度铺在含有100 mg l-1卡那霉素的M9平板上,并在30°C培养。通过使用引物对spmA-Sall和pK18mob-269, GCTTCCCAACCTTACCAGAG进行PCR分析测试Kan抗性转接合子。
构建含有互补spmABC基因的质粒pME6032-spm100
使用表S5中的引物从恶臭假单胞菌S16的总DNA中扩增包含spmABC基因编码区和spmA ATG上游100 bp的4.45 kb片段,然后克隆到pME6032[32]中,生成重组质粒pME6032-spm100。
构建恶臭假单胞菌S16的mfs、sapd、pnao、nicA2基因破坏突变体
通过PCR从恶臭假单胞菌S16的总DNA中扩增mfs、sapd、pnao和nicA2的DNA片段,并克隆到pK18mob的多克隆位点中。PCR引物对序列如表S6所示。通过电转化转化恶臭假单胞菌S16,条件如下:将0.5-1μg DNA加入100μl恶臭假单胞菌S16的电感受态细胞中,使用Bio-Rad Gene-Pulser Xcell(Bio-Rad Laboratories, Hercules, CA)在12 kV cm-1、200 Ω、25μF下电击。
相关新闻推荐
1、低浓度微酸性电解水对纯培养及海虾表面接种菌的杀菌效果以及作用机制(三)