结果
hk11和rr11缺失突变体的遗传确认。
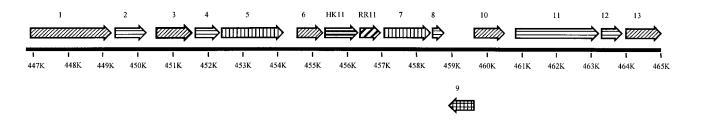
通过使用blastP算法将相邻开放阅读框的推导氨基酸序列与GenBank数据库进行比较,对hk/rr11区域周围的遗传位点进行了注释。该位点的图谱和描述如图1所示。hk11基因位于变形链球菌基因组数据库中的bp 455345至456349处,编码一个推定的334个氨基酸的假设蛋白,预测分子量为38,113 Da。hk11 ORF与来自化脓性链球菌的推定的双组分传感器组氨酸激酶(登录号AAK34394)(blast相似性得分=352);来自肺炎链球菌的推定的组氨酸激酶HK03(CAB54570)(blast得分=244);以及来自耐盐芽孢杆菌的推定的组氨酸激酶BH1199(BAB04918)(blast得分=177)具有最高相似性。
编码rr11的ORF位于bp 456336至456983,编码一个推定的215个氨基酸的假设蛋白,预测分子量为24,067 Da。有趣的是,这些基因重叠了12个核苷酸,并且应答调节蛋白编码基因在其推定起始密码子5'端有一个启动子样结构。在这个5'近端区域,-18至-10序列TACCAACT与肺炎链球菌的com-box共识序列仅相差一个碱基对(TACGAACT)。这些com-box基因构成了CSP介导的调节子的一部分。hk11基因也有推定的-10(-12,TAATGA)和-35(TGTTATGGA)启动子序列。然而,它在这个附近似乎没有com-box。与hk11基因类似,rr11与来自化脓性链球菌的相应应答调节基因(SPy1621)和来自肺炎链球菌的R03基因具有最高同源性,但与来自枯草芽孢杆菌的假设应答调节基因yvqC具有更高的相似性,而不是与耐盐芽孢杆菌的同源物。目前尚未将任何推定的底物或信号分配给这些生物体中的任何这些系统。
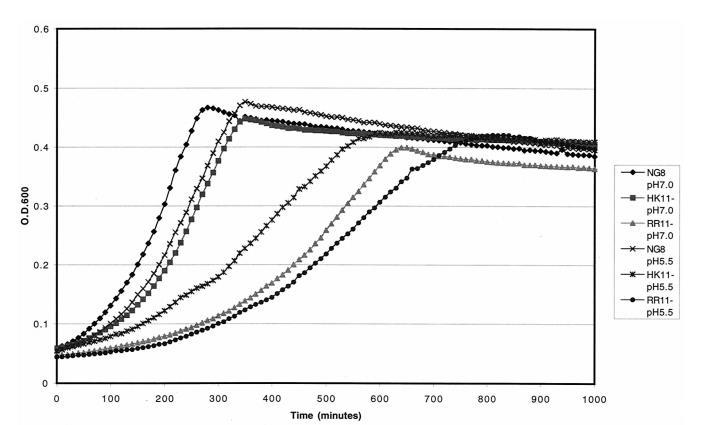
PCR确认证明,hk11和rr11突变体的靶基因在其各自的突变体中正确地被Erm盒替换(数据未显示)。经PCR确认的突变体分别命名为SMHK11和SMRR11。生长动力学显示,两种突变体在SDM培养基中的生长速率均下降(倍增时间增加):SMHK11的Td=1.47小时,SMRR11的Td=1.43小时,而亲本菌株的Td=1.27小时。然而,突变体在TYEG(图2)或SDM培养基(数据未显示)中生长12小时后的最终生长产量似乎与亲本菌株相同。两种突变体与亲本菌株类似,能够变得具有遗传感受态,并且无论是否添加CSP,都能被质粒DNA转化,这表明由hk/rr11编码的系统不影响该生物的感受态发展。
删除hk11或rr11导致生物膜形成缺陷。
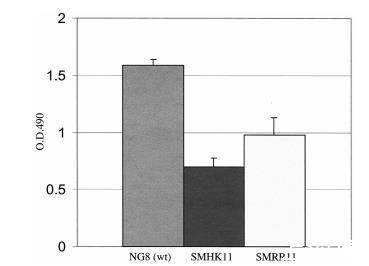
删除hk11或rr11基因导致生物膜形成缺陷,如图3所示。菌株SMHK11的生物膜密度约为亲本菌株NG8的50%,菌株SMRR11的密度约为亲本菌株的75%。观察到的突变体生物膜密度降低不太可能是由于它们略微降低的生长速率所致,特别是因为它们在中性pH下的生长产量与亲本菌株在TYEG(图2)和SDM培养基(数据未显示)中几乎相同。
通过SEM更仔细地检查生物膜发现,突变体形成的生物膜外观与亲本生物膜非常不同。突变体的生物膜似乎具有海绵状结构,其中似乎存在大的细胞间间隙(图4)。我们发现,在生物膜测定制备过程中,由hk11和rr11突变体形成的这种生物膜比亲本菌株形成的生物膜更容易从表面洗脱。此外,突变体的静息细胞粘附到粘蛋白包被的聚苯乙烯表面的能力降低(粘附到表面的细胞百分比±[标准差]:NG8,12.08[2.04];HK11,6.77[1.32];RR11,8.36[1.53])。综上所述,海绵状结构的明显缺陷以及细胞对表面粘附的较低亲和力可能导致观察到的突变体生物量减少。SEM还显示,当以生物膜形式生长时,SMHK11和SMRR11都形成了非常长的链,与野生型菌株相比。

图4.扫描电子显微照片显示变形链球菌菌株形成的生物膜的空间分布和结构。
由于怀疑这种表型与CSP激活的途径有关,我们检查了CSP对hk/rr11突变体细胞链形成的影响。向生物膜培养物中添加CSP并未显著改变SMHK11和SMRR11突变体形成的链长度。为了从SEM图像定量链长度,从随机选择的四个独立链获得平均值。每个链的平均细胞数(±SD)如下:NG8(野生型),17(8.04);HK11,42(13.8);RR11,38(11.3)。
hk11突变体存在酸耐受性缺陷。
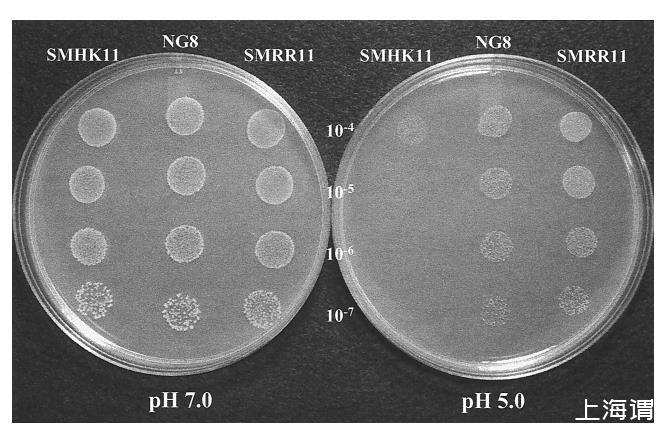
与亲本菌株NG8相比,SMHK11和SMRR11突变体在pH 5.5的液体培养物中均显示出生长速率降低。NG8的Td为100±1分钟。SMHK11的Td为178±3分钟,而SMRR11每207±9分钟倍增一次(图2)。突变体SMHK11在pH 5.0的琼脂平板上的生长也大大减弱,尽管它在pH 7.0的平板上(图5)和pH 7.0的肉汤中(图2)生长与亲本菌株一样好。有趣的是,我们未能在pH 5.0平板上检测到SMRR11突变体与亲本菌株NG8生长之间的差异。为了更仔细地确定删除hk11或rr11基因是否影响可诱导的ATR,我们使用先前描述的方法测定了在液体培养物中生长的突变体的对数期ATR。结果显示,SMHK11突变体相对于亲本菌株NG8具有降低的ATR(图6)。然而,删除rr11仅导致可诱导ATR轻微下降,如菌株SMRR11所示。
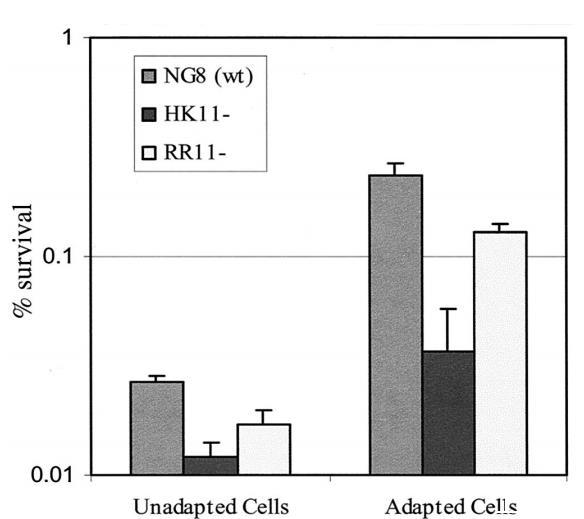
SMHK11的酸刺激子。通过比较暴露于pH 7.5和5.5条件下30分钟的总细胞标记蛋白的二维凝胶电泳,分析了NG8和SMHK11的ATR背后蛋白质表达的变化。在这些实验中监测的594个蛋白质中,所有那些在酸暴露30分钟后表达差异>2.0或<0.5的蛋白质被分别分类为属于NG8和SMHK11的酸刺激子。NG8的酸刺激子包括19个蛋白质,其中12个显示表达增加,7个显示表达减少(图7A和B;表3)。有趣的是,在酸冲击后显示表达增加的12个NG8蛋白质中,有四个蛋白质在突变菌株中没有被诱导(图7C和D;表3)。这些结果证实,SMHK11在诱导酸应激蛋白方面相对于亲本菌株NG8存在缺陷,并证实hk11在可诱导的ATR中起着重要作用。
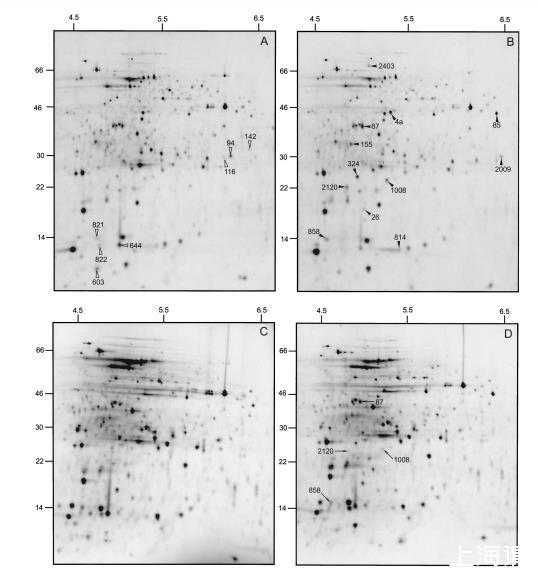
表3.NG8和SMHK11中诱导或抑制的蛋白质
| 因酸应激表达变化>3倍的蛋白质(NG8) | 是否属于SMHK11酸应激共有 |
| 表达增加 | |
| 4a(丙酮酸脱氢酶) | 是 |
| 26(未知) | 是 |
| 85(半胱氨酸合酶) | 是 |
| 87(外切多磷酸酶) | 否 |
| 155(未知) | 是 |
| 324(未知) | 是 |
| 814(未知) | 是 |
| 858(未知) | 否 |
| 1008(组氨酸激酶候选物) | 否 |
| 2009(未知) | 是 |
| 2120(未知) | 否 |
| 2403(未知) | 是 |
| 表达减少b | |
| 94(未知) | 是 |
| 116(未知) | 是 |
| 142(未知) | 是 |
| 603(未知) | 是 |
| 644(未知) | 是 |
| 821(未知) | 是 |
| 822(未知) | 是 |
相关新闻推荐
1、加勒比海红树林中发现一种长达2厘米的巨型细菌,拥有庞大的基因组!
2、生长曲线分析仪监测突变体在抗生素利福平处理后的生长恢复情况(一)