实时定量PCR分析在7,500实时PCR系统(Applied Biosystems)上进行。每个反应在包含以下组分的反应混合物中进行:1x浓缩的商业缓冲液(随聚合酶提供,Metabion)、0.625 U Taq聚合酶(Metabion)、4 mM MgCl?、30,000倍稀释的SYBR Green(Sigma)、5% DMSO、0.5 ng/μl乙酰化BSA(Sigma)、0.8%甘油(ROTH)、400μM dNTP混合物(Metabion)、前向和后向特异性引物(各400μM)、cDNA模板和水,最终体积为25μl。反应进行初始变性步骤(95°C 3分钟),随后进行45个循环的变性(95°C 15秒)和引物退火-延伸(60°C 1分钟)。
每个循环的退火-延伸步骤读取荧光。循环后,在60至95°C范围内以0.33°C的增量进行熔解温度分析。在每个实验中,自动调整背景范围,并手动调整Ct评估的阈值。对于每个cDNA样品,使用三种模板量进行六次反应,每种重复两次。对于每个研究的基因,单独选择cDNA量(如果可能,所有基因相同)以获得在14至34个循环范围内的Ct值。基于三个cDNA量之间的预期Ct差异以及产物熔解曲线评估结果质量。在计算中省略罕见的异常结果。通过使用乳酸乳球菌参考基因tuf和purM(分别编码延伸因子TU和磷酸核糖氨基咪唑合成酶)对结果进行标准化。使用Primer Express软件(Applied Biosystems)设计每个基因的特异性引物(表1)。在使用前,验证引物的PCR反应效率相等。将所得数据从qPCR循环仪程序导出的文件进行处理,并导入Excel表格中以便于计算靶基因与参考基因之间的表达比率。这些比率通过△Ct方法计算,在每个实验中分别使用参考基因Ct的几何平均值。
碳源利用的表型测试 通过生长测试和发酵模式分析研究突变细胞与亲本菌株相比的表型变化。
生长测试在Microbiology Reader Analyser, Bioscreen C (Labsystems)上进行,使用0.2 ml补充有所需糖的CDM。在长达100小时的生长过程中,每30分钟记录一次细菌培养物OD600的变化。
使用API 50CH测试(按照制造商的说明(BioMérieux))测定49种糖的发酵模式,并在30°C有氧或厌氧条件下培养12、24和36小时后记录。
通过表型微阵列系统(Biolog,USA)根据制造商的说明进一步全局测量代谢谱。将乳酸乳球菌菌株划线在含有G-M17琼脂的平板上。从平板上刮下菌落,并滴定到IF-0a接种液(Biolog)中,添加生长补充剂和Biolog氧化还原四唑鎓染料,直到溶液达到所需的透光度,根据Biolog为链球菌物种推荐的标准方案。将100微升等分试样添加到碳源板(PM1和PM2)的每个孔中。将板在30°C下在有氧OmniLog培养箱板阅读器中培养,并通过测定四唑鎓染料的比色还原动力学测量代谢活性。表型微阵列使用Biolog的氧化还原测定法,利用细胞呼吸或发酵作为通用报告基因,并提供表型的精确量化。如果孔中的表型强烈"阳性",则细胞代谢活跃,还原四唑鎓染料并形成强烈的颜色。如果它们的代谢活性减慢或停止,则形成较少的颜色或不形成颜色。数据在大约72小时内每10分钟收集一次。这段时间足以使阳性对照孔中显色,而阴性对照孔保持无色。使用Biolog动力学和参数软件分析数据。PM1和PM2 Biolog测定评估细菌利用测定中使用的190种碳化合物中任何一种作为唯一碳源的能力。
结果
包含claR基因的DNA区域的结构特征 图1描绘了来自乳酸乳球菌IL1403的claR基因(原yebF)的染色体区域。ClaR显示出与RpiR家族转录调控因子的高度序列相似性:它是一种双结构域蛋白,具有67个残基的N端DNA结合螺旋-转角-螺旋(HTH)基序和123个残基的C端糖异构酶(SIS)结构域(图1a;http://pfam.sanger.ac.uk/)。
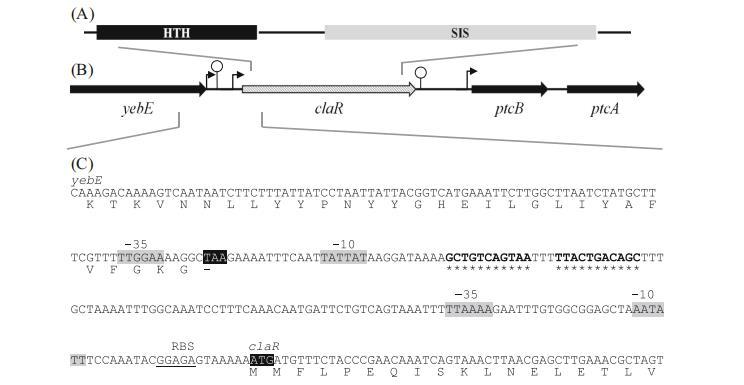
claR上游是yebE基因,编码一种假设蛋白。在这两个基因之间的基因间区域内,定位了一个推定rho非依赖型终止子,其自由能值(dG)为-15 kcal/mol(图1b,c)。为了验证其功能性,将该终止子克隆到pGBT58载体中,位于启动子和编码儿茶酚2,3-双加氧酶的xylE基因之间。在携带该构建体的大肠杆菌菌株中,获得的儿茶酚2,3-双加氧酶活性水平达到0.50±0.04 U,低于观察到的对照值(不含yebE终止子的pGBT58)1.25±0.16 U。因此,与携带野生型pGBT58载体的菌株相比,yebE终止子的存在导致儿茶酚加氧酶活性降低60%。
根据计算机分析,claR基因前面至少有兩個推定启动子(图1b,c),表明存在多个潜在的转录起始位点。鉴定出的claR上游潜在-10和-35启动子区域均未与分别定义为TATAAT和TTGACA的启动子共有序列完全一致。这些潜在启动子之一位于yebE rho非依赖型终止子上游(图1b,c)。为了验证在claR上游DNA区域是否存在功能性启动子,将包含上述推定启动子的DNA片段插入到启动子探针pJIM2374载体中的luxAB报告基因前面,并导入乳酸乳球菌LL302。当测试claR上游区域时,检测到显著的荧光素酶活性,表明其中至少嵌入了一个功能性启动子。
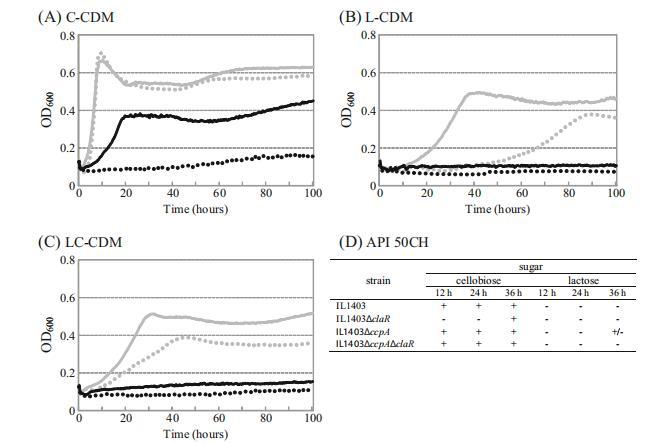
claR缺失突变体的表型 在我们先前的研究中,利用pGhost9::ISS1对发酵乳糖的ccpA突变体进行随机诱变。通过此程序,在补充有X-Gal的平板上,我们分离了几個在ccpA突变体背景中具有β-半乳糖苷酶阴性表型的突变体。其中,在ccpA突变体中claR(yebF)的失活导致其丧失乳酸发酵能力。因此,在本研究中,在IL1403野生型菌株和IL1403△ccpA中均进行了claR的缺失,分别产生IL1403△claR和IL1403△ccpA△claR突变体。随后,测试了IL1403△claR和IL1403△ccpA△claR在补充有各种糖的CDM中的生长,并与它们各自的亲本菌株进行了比较。在C-CDM中,IL1403△claR突变体最初显示无生长能力,并且在长时间培养后观察到细胞光密度的微小增加(图2a)。在IL1403△claR菌株中缺失ccpA导致所得IL1403△ccpA△claR菌株在C-CDM中的生长部分恢复(图2a)。与它们的亲本IL1403和IL1403△ccpA菌株相比,IL1403△claR和IL1403△ccpA△claR在L-CDM中均无生长(图2b)。向该培养基中添加诱导浓度的纤维二糖(LC-CDM)并未恢复突变体的生长(图2c)。在IL1403和IL1403△ccpA菌株中缺失claR基因对突变体在含熊果苷、七叶苷、半乳糖、葡萄糖或水杨苷的培养基中的生长没有影响(数据未显示)。
相关新闻推荐
1、砷氧化菌株在砷胁迫中生长曲线测定及重金属铜的耐受性(一)
3、类肠膜魏斯氏菌对猪生长性能、粪菌和代谢产物的影响——摘要、材料与方法