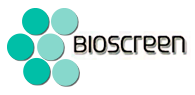一、未培养微生物的困境与破局
微生物生态学研究长期面临一个核心矛盾:自然界中绝大多数微生物无法在实验室培养。传统方法需反复尝试温度、pH、盐度等条件组合,耗时数月甚至数年。更棘手的是,基于功能基因注释的预测对生长条件束手无策——氧气耐受性、最适温度等参数难以通过代谢路径反推。
GenomeSPOT的诞生直击这一痛点。它绕过基因注释的局限,直接从氨基酸组成频率中挖掘生长信号,为未培养微生物的“解锁”提供了新钥匙。
二、氨基酸频率:隐藏的生长密码
1.颠覆性思路:从序列到环境的直接映射
传统基因组分析依赖功能注释,但GenomeSPOT发现:微生物为适应环境会调整蛋白质的氨基酸组成。例如,嗜盐菌的膜蛋白富含带电氨基酸以维持结构稳定性;耐热菌倾向使用疏水性氨基酸防止高温变性。这种适应性进化在序列上留下可量化的痕迹。
2.局部化加权:提升预测精度的关键
研究团队发现,不同亚细胞定位的蛋白质对环境的响应程度不同。膜蛋白的氨基酸组成对盐度更敏感,而胞内蛋白则与pH关联更强。通过分离计算胞内、胞外、膜蛋白的氨基酸频率,模型对pH的预测R²提升0.36,显著突破基线瓶颈。
三、技术内核:数据驱动的统计模型
1.训练数据与特征工程
模型基于15,596株已培养微生物的基因组训练,涵盖细菌和古菌。每个基因组的输入特征是其全部蛋白质的20种氨基酸相对频率(或分亚细胞定位的频率矩阵)。输出目标包括四类生长参数:
氧气耐受性(分类模型):厌氧/兼性/好氧
温度、盐度、pH(回归模型):最适值及耐受范围
2.模型选择与性能验证
氧气耐受性预测平衡准确率达92%,仅需两种关键氨基酸即可实现88%的准确率;
最适温度预测R²=0.73,盐度R²=0.81,pH经亚细胞优化后R²显著提升;
交叉验证证明:即使基因组完整度仅10%(模拟MAGs片段化),预测误差仍在可控范围。
四、实战性能:85,205个基因组的检验
在GTDB数据库的大规模测试中,GenomeSPOT展现出极强鲁棒性:
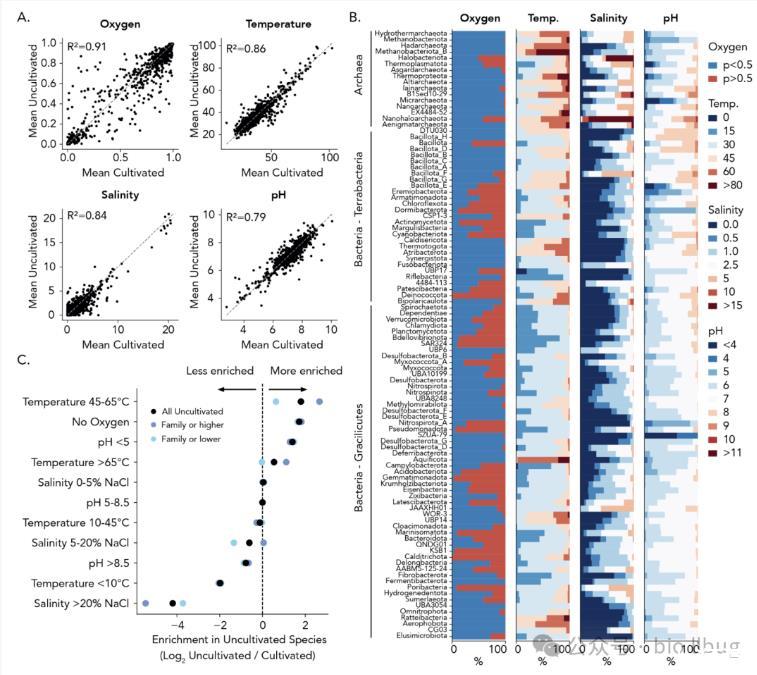
覆盖率:仅0.3%基因组因蛋白数量不足(<700个)无法预测;
速度:单基因组预测仅需5-10秒;
未培养微生物的新发现:预测显示,未培养物种更倾向极端环境(如高温厌氧、高酸),暗示实验室培养条件的设计需突破常规思路。
案例启示:同一环境样本中的MAGs预测出差异显著的生长条件,揭示微生物群落通过生态位分化实现共存——这是传统纯培养实验难以捕捉的细节。
五、操作指南:从安装到结果解读
1.环境配置的雷区
Python版本必须为3.8.16–3.11,scikit-learn==1.2.2版本错误将导致模型崩溃;
推荐使用conda创建独立环境,避免依赖冲突。
2.输入文件的灵活处理
若仅有基因组序列(.fna),需先用Prodigal预测蛋白序列(.faa);
支持压缩文件直接输入,减少存储压力。
3.结果表格的深度解读
输出文件包含五列关键信息:
value:氧气耐受性返回"tolerant"或"not tolerant",其他为数值;
error:连续变量为RMSE,氧气耐受性为置信概率(>0.75可信度高);
is_novel:标记基因组特征与训练集差异>98%,结果需审慎参考;
warning:如"min_exceeded"提示预测值超合理范围(如盐度<0被自动修正)。
批量处理技巧:使用Shell并行命令控制任务数(如同时运行10个基因组),结合join_outputs脚本一键合并TSV结果表。
六、局限与未来:工具如何推动微生物学
当前瓶颈
pH预测精度仍落后于温度/盐度;
极端环境微生物(如超嗜热菌)的训练样本不足,预测存在偏差;
氨基酸组成与环境适应的因果机制尚未完全明确。
应用场景拓展
定向培养:为未培养微生物定制培养基(如根据预测pH调整缓冲体系);
生物修复:快速筛选耐重金属或降解污染物的菌株;
工业菌株开发:预选耐高温蛋白酶生产菌,缩短发酵工艺优化周期。
七、结语:从“黑箱”到“解码器”
GenomeSPOT的价值不仅在于预测精度,更在于其颠覆了生长条件研究的范式。它将不可培养微生物从“生态学谜题”转化为可量化的数据对象,为微生物资源挖掘按下加速键。随着训练数据的扩充和模型迭代,未来或可实现对更多参数(如重金属耐受性)的预测——这将是微生物学走向“可预测性科学”的关键一步。
相关新闻推荐
1、微生物的分离培养方法、操作步骤及注意事项——划线分离法、涂布平板法